Existe-t-il des différences d’immunité face au coronavirus SARS-CoV-2 entre populations de diverses régions géographiques? Une partie de la réponse est à rechercher dans les génomes de ces groupes de personnes et, plus particulièrement, dans les gènes HLA, responsables du système immunitaire dit adaptatif. Ces gènes ont la particularité de différer souvent entre individus. Des milliers de variants (ou allèles) possibles ont été identifiés et tous ne possèdent pas la même efficacité pour lutter contre un nouveau virus. La fréquence de ces allèles varie d’une population à l’autre du fait des migrations passées et de leur adaptation à divers environnements.
Des scientifiques de l’Université de Genève (UNIGE), en collaboration avec l’Institut Max Planck de Jéna (Allemagne) et l’Université d’Adélaïde (Australie), identifient les variants HLA potentiellement les plus efficaces contre 7 virus, dont le nouveau coronavirus, et mettent en lumière des variations significatives entre populations.
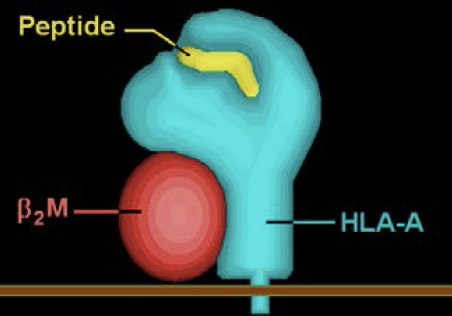
Les gènes du système HLA (Human Leukocyte Antigen, ou antigènes des leucocytes humains) produisent des molécules HLA qui se placent à la surface des cellules. Lorsqu’un virus infecte un organisme, les protéines de l’envahisseur sont d’abord découpées en petits fragments appelés peptides. Les molécules HLA se lient ensuite à ces fragments et les exposent à la surface des cellules, déclenchant ainsi une cascade de réactions immunitaires destinées à éliminer le virus.
À l’aide d’outils bioinformatiques et de modèles statistiques, les chercheurs ont tenté d’identifier, parmi les quelques 450 molécules HLA les plus communes dans des centaines de populations à travers le monde, celles qui se lient le plus fortement aux peptides du nouveau coronavirus. Plus de 7000 peptides peuvent être dérivés de l’ensemble des protéines virales du coronavirus.
En classant les quelques 450 molécules HLA selon leur capacité plus ou moins forte à lier les peptides du coronavirus, l’étude fournit un inventaire de référence essentiel pour identifier les susceptibilités ou résistances génétiques plus ou moins grandes des individus au virus. Elle montre également que la fréquence de ces variants HLA diffère de manière significative d’une population à l’autre.
Dans la même étude, les auteurs ont analysé les liaisons HLApeptides pour la totalité des protéines de 6 autres virus à potentiel pandémique (2 autres coronavirus, 3 virus influenza de la grippe et le virus HIV-1 du SIDA). Il en ressort que de nombreux variants HLA sont capables de se lier fortement aux peptides de l’ensemble des 7 virus étudiés. D’autres font de même pour tous les virus de type respiratoire (coronavirus et influenza). Cela signifie donc qu’il existe de nombreuses molécules HLA «généralistes», c’est-à-dire qui sont efficaces contre de nombreux virus différents.
Une suite logique à cette étude sera de déterminer quels sont précisément les peptides du coronavirus qui sont le plus fortement liés aux molécules HLA. Ce sont en effet ces peptides qui auront les plus fortes chances de déclencher une réaction immunitaire efficace. Leur identification sera une information essentielle pour le développement d’un vaccin.


 Existe-t-il des différences d’immunité face au coronavirus SARS-CoV-2 entre populations de diverses régions géographiques? Une partie de la réponse est à rechercher dans les génomes de ces groupes de personnes et, plus particulièrement, dans les gènes HLA, responsables du système immunitaire dit adaptatif. Une étude de l'Université de Genève fait la comparaison et identifie les variants génétiques potentiellement les plus efficaces. Dans un communiqué de presse, Alicia Sanchez-Mazas, professeure à l'Unité d’Anthropologie du Département de génétique et évolution de Genève, explique ces résultats.
Existe-t-il des différences d’immunité face au coronavirus SARS-CoV-2 entre populations de diverses régions géographiques? Une partie de la réponse est à rechercher dans les génomes de ces groupes de personnes et, plus particulièrement, dans les gènes HLA, responsables du système immunitaire dit adaptatif. Une étude de l'Université de Genève fait la comparaison et identifie les variants génétiques potentiellement les plus efficaces. Dans un communiqué de presse, Alicia Sanchez-Mazas, professeure à l'Unité d’Anthropologie du Département de génétique et évolution de Genève, explique ces résultats.